Expression Evaluation via eval() (Experimental)
版本0.13中的新功能。
顶层函数pandas.eval()实现Series和DataFrame对象的表达式求值。
注意
要受益于使用eval(),您需要安装numexpr。有关详细信息,请参阅recommended dependencies section。
使用eval()来表达式求值而不是纯Python是两个方面:1)大的DataFrame对象被更有效地计算,2)大的算术和布尔表达式由底层引擎一次性计算(默认情况下,numexpr用于计算)。
注意
对于简单表达式或涉及小型DataFrames的表达式,不应使用eval()。事实上,对于较小的表达式/对象,eval()比纯粹的Python要慢许多个数量级。一个好的经验法则是,当您拥有超过10,000行的DataFrame时,只使用eval()。
eval()支持引擎支持的所有算术表达式,除了一些仅在pandas中可用的扩展。
注意
帧越大,表达式越大,使用eval()可以看到的加速越快。
Supported Syntax
这些操作由pandas.eval()支持:
- Arithmetic operations except for the left shift (
<<) and right shift (>>) operators, e.g.,df + 2 * pi / s ** 4 % 42 - the_golden_ratio - 比较操作,包括链式比较,例如
2 df df2 - Boolean operations, e.g.,
df < df2 and df3 < df4 or not df_bool listandtupleliterals, e.g.,[1, 2]or(1, 2)- 属性访问权限,例如
df.a - 下标表达式,例如
df[0] - 简单变量评估,例如
pd.eval('df')(这不是很有用) - Math functions, sin, cos, exp, log, expm1, log1p, sqrt, sinh, cosh, tanh, arcsin, arccos, arctan, arccosh, arcsinh, arctanh, abs and arctan2.
此Python语法为不允许:
eval() Examples
pandas.eval()适用于包含大型数组的表达式。
首先,让我们创建一些大小合适的数组:
In [13]: nrows, ncols = 20000, 100
In [14]: df1, df2, df3, df4 = [pd.DataFrame(np.random.randn(nrows, ncols)) for _ in range(4)]
现在让我们比较使用纯粹的Python和eval()将它们添加在一起:
In [15]: %timeit df1 + df2 + df3 + df4
10 loops, best of 3: 24.6 ms per loop
In [16]: %timeit pd.eval('df1 + df2 + df3 + df4')
100 loops, best of 3: 8.36 ms per loop
现在让我们做同样的事情,但比较:
In [17]: %timeit (df1 > 0) & (df2 > 0) & (df3 > 0) & (df4 > 0)
10 loops, best of 3: 30.9 ms per loop
In [18]: %timeit pd.eval('(df1 > 0) & (df2 > 0) & (df3 > 0) & (df4 > 0)')
100 loops, best of 3: 16.4 ms per loop
eval()也可以使用未对齐的pandas对象:
In [19]: s = pd.Series(np.random.randn(50))
In [20]: %timeit df1 + df2 + df3 + df4 + s
10 loops, best of 3: 38.4 ms per loop
In [21]: %timeit pd.eval('df1 + df2 + df3 + df4 + s')
100 loops, best of 3: 9.31 ms per loop
注意
操作如
1 and 2 # would parse to 1 & 2, but should evaluate to 2 3 or 4 # would parse to 3 | 4, but should evaluate to 3 ~1 # this is okay, but slower when using eval
应该在Python中执行。如果尝试使用非类型为bool或np.bool_的标量操作数执行任何布尔/逐位运算,则会引发异常。同样,你应该在纯Python中执行这些类型的操作。
The DataFrame.eval method (Experimental)
版本0.13中的新功能。
除了顶层pandas.eval()函数,您还可以评估DataFrame的“上下文”中的表达式。
In [22]: df = pd.DataFrame(np.random.randn(5, 2), columns=['a', 'b'])
In [23]: df.eval('a + b')
Out[23]:
0 -0.246747
1 0.867786
2 -1.626063
3 -1.134978
4 -1.027798
dtype: float64
作为有效pandas.eval()表达式的任何表达式也是有效的DataFrame.eval()表达式,还有一个好处,到您想要评估的列的DataFrame的名称。
此外,您可以在表达式中执行列的分配。这允许公式计算。分配目标可以是新的列名称或现有的列名称,它必须是有效的Python标识符。
版本0.18.0中的新功能。
inplace关键字确定此分配是否对原始DataFrame执行,或返回带有新列的副本。
警告
对于向后兼容性,如果未指定,inplace默认为True。这将在未来版本的pandas中改变 - 如果你的代码依赖于一个内部赋值,你应该更新来显式设置inplace=True
In [24]: df = pd.DataFrame(dict(a=range(5), b=range(5, 10)))
In [25]: df.eval('c = a + b', inplace=True)
In [26]: df.eval('d = a + b + c', inplace=True)
In [27]: df.eval('a = 1', inplace=True)
In [28]: df
Out[28]:
a b c d
0 1 5 5 10
1 1 6 7 14
2 1 7 9 18
3 1 8 11 22
4 1 9 13 26
当inplace设置为False时,将返回带有新列或已修改列的DataFrame的副本,原始帧不变。
In [29]: df
Out[29]:
a b c d
0 1 5 5 10
1 1 6 7 14
2 1 7 9 18
3 1 8 11 22
4 1 9 13 26
In [30]: df.eval('e = a - c', inplace=False)
Out[30]:
a b c d e
0 1 5 5 10 -4
1 1 6 7 14 -6
2 1 7 9 18 -8
3 1 8 11 22 -10
4 1 9 13 26 -12
In [31]: df
Out[31]:
a b c d
0 1 5 5 10
1 1 6 7 14
2 1 7 9 18
3 1 8 11 22
4 1 9 13 26
版本0.18.0中的新功能。
为了方便,可以通过使用多行字符串来执行多个分配。
In [32]: df.eval("""
....: c = a + b
....: d = a + b + c
....: a = 1""", inplace=False)
....:
Out[32]:
a b c d
0 1 5 6 12
1 1 6 7 14
2 1 7 8 16
3 1 8 9 18
4 1 9 10 20
在标准Python中的等价将是
In [33]: df = pd.DataFrame(dict(a=range(5), b=range(5, 10)))
In [34]: df['c'] = df.a + df.b
In [35]: df['d'] = df.a + df.b + df.c
In [36]: df['a'] = 1
In [37]: df
Out[37]:
a b c d
0 1 5 5 10
1 1 6 7 14
2 1 7 9 18
3 1 8 11 22
4 1 9 13 26
版本0.18.0中的新功能。
query方法获得了inplace关键字,该关键字确定查询是否修改原始帧。
In [38]: df = pd.DataFrame(dict(a=range(5), b=range(5, 10)))
In [39]: df.query('a > 2')
Out[39]:
a b
3 3 8
4 4 9
In [40]: df.query('a > 2', inplace=True)
In [41]: df
Out[41]:
a b
3 3 8
4 4 9
警告
Unlike with eval, the default value for inplace for query is False. 这与以前版本的熊猫一致。
Local Variables
在pandas版本0.14中,本地变量API已更改。在pandas 0.13.x中,你可以像在标准Python中一样引用局部变量。例如,
df = pd.DataFrame(np.random.randn(5, 2), columns=['a', 'b'])
newcol = np.random.randn(len(df))
df.eval('b + newcol')
UndefinedVariableError: name 'newcol' is not defined
从生成的异常中可以看出,不再允许使用此语法。您必须通过将@字符放在名称前,显式引用要在表达式中使用的任何局部变量。例如,
In [42]: df = pd.DataFrame(np.random.randn(5, 2), columns=list('ab'))
In [43]: newcol = np.random.randn(len(df))
In [44]: df.eval('b + @newcol')
Out[44]:
0 -0.173926
1 2.493083
2 -0.881831
3 -0.691045
4 1.334703
dtype: float64
In [45]: df.query('b < @newcol')
Out[45]:
a b
0 0.863987 -0.115998
2 -2.621419 -1.297879
如果你不用局部变量前缀@,pandas将引发一个异常告诉你该变量是未定义的。
当使用DataFrame.eval()和DataFrame.query()时,这允许您有一个局部变量和一个DataFrame表达式中的名称。
In [46]: a = np.random.randn()
In [47]: df.query('@a < a')
Out[47]:
a b
0 0.863987 -0.115998
In [48]: df.loc[a < df.a] # same as the previous expression
Out[48]:
a b
0 0.863987 -0.115998
With pandas.eval() you cannot use the @ prefix at all, because it isn’t defined in that context. 如果您尝试在对pandas.eval()的顶级调用中尝试使用@,则pandas会让您知道这一点。例如,
In [49]: a, b = 1, 2
In [50]: pd.eval('@a + b')
File "<string>", line unknown
SyntaxError: The '@' prefix is not allowed in top-level eval calls,
please refer to your variables by name without the '@' prefix
在这种情况下,你应该像在标准Python中那样引用变量。
In [51]: pd.eval('a + b')
Out[51]: 3
pandas.eval() Parsers
有两个不同的解析器和两个不同的引擎可以用作后端。
默认的'pandas'解析器允许更直观的语法来表达类查询操作(比较,连接和析取)。特别地,使&和|运算符的优先级等于相应的布尔运算and和or。
例如,上述连接可以不用括号写。或者,您可以使用'python'解析器强制执行严格的Python语义。
In [52]: expr = '(df1 > 0) & (df2 > 0) & (df3 > 0) & (df4 > 0)'
In [53]: x = pd.eval(expr, parser='python')
In [54]: expr_no_parens = 'df1 > 0 & df2 > 0 & df3 > 0 & df4 > 0'
In [55]: y = pd.eval(expr_no_parens, parser='pandas')
In [56]: np.all(x == y)
Out[56]: True
相同的表达式可以与字and一起被“anded”:
In [57]: expr = '(df1 > 0) & (df2 > 0) & (df3 > 0) & (df4 > 0)'
In [58]: x = pd.eval(expr, parser='python')
In [59]: expr_with_ands = 'df1 > 0 and df2 > 0 and df3 > 0 and df4 > 0'
In [60]: y = pd.eval(expr_with_ands, parser='pandas')
In [61]: np.all(x == y)
Out[61]: True
这里的and和or运算符具有与在vanilla Python中相同的优先级。
pandas.eval() Backends
还有一个选项让eval()操作与纯粹的Python相同。
注意
使用'python'引擎通常不有用,除了测试其他评估引擎。您将使用eval()和engine='python'实现no性能优势,实际上可能会造成性能损失。
你可以通过使用pandas.eval()和'python'引擎来看到这一点。它比在Python中评估同一个表达式慢一点(不是太多)
In [62]: %timeit df1 + df2 + df3 + df4
10 loops, best of 3: 24.2 ms per loop
In [63]: %timeit pd.eval('df1 + df2 + df3 + df4', engine='python')
10 loops, best of 3: 25.2 ms per loop
pandas.eval() Performance
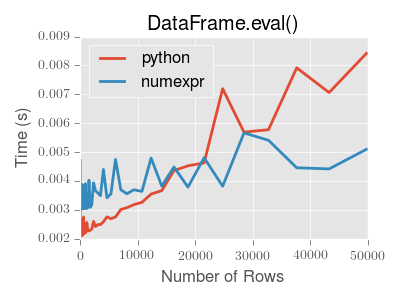
eval()旨在加速某些类型的操作。特别地,涉及具有大的DataFrame / Series对象的复杂表达式的那些操作应当看到显着的性能益处。这里是一个图表,显示pandas.eval()的运行时间作为计算中涉及的框架大小的函数。这两条线是两个不同的引擎。
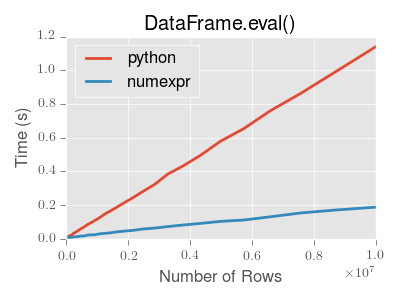
注意
使用纯Python,较小对象(大约15k-20k行)的操作速度更快:
此图使用DataFrame创建,每个列包含使用numpy.random.randn()生成的浮点值。
Technical Minutia Regarding Expression Evaluation
必须在Python空间中评估导致对象dtype或涉及datetime操作(因为NaT)的表达式。此行为的主要原因是保持与numpy版本的向后兼容性在numpy的这些版本中,对ndarray.astype(str)的调用将截断长度超过60个字符的任何字符串。第二,我们不能将object数组传递到numexpr,因此字符串比较必须在Python空间中求值。
结果是,这仅适用于object-dtype的表达式。所以,如果你有一个表达式 - 例如
In [64]: df = pd.DataFrame({'strings': np.repeat(list('cba'), 3),
....: 'nums': np.repeat(range(3), 3)})
....:
In [65]: df
Out[65]:
nums strings
0 0 c
1 0 c
2 0 c
3 1 b
4 1 b
5 1 b
6 2 a
7 2 a
8 2 a
In [66]: df.query('strings == "a" and nums == 1')
Out[66]:
Empty DataFrame
Columns: [nums, strings]
Index: []
比较的数字部分(nums == 1)将由numexpr
In general, DataFrame.query()/pandas.eval() will evaluate the subexpressions that can be evaluated by numexpr and those that must be evaluated in Python space transparently to the user. 这是通过从其参数和运算符推断表达式的结果类型来完成的。